Pymatgenによる化合物データの取得(Materials Project)
本記事では、Pymatgenによる化合物データ取得方法を紹介していく。
Pythonの基礎を習得していることを前提として話を進めていく。
目次:
Pymatgenとは
pymatgenとは、材料解析のためのオープンソースのpythonライブラリであり、以下の特徴があげられる。
原子や分子の構造をPythonのクラスで柔軟に表現
- VASPやGaussianといった多くのフォーマットに対応
- 位相図の作成や拡散解析などが可能な強力な解析ツール
- 状態密度やバンド構造など、電子構造解析も可能
- 結晶学のオープンデータベースであるMaterials Project REST APIと連動(今回はここ)
データベースにアクセスするためには、API KEYの取得が必要になる。
以下から、登録が可能である。
materialsproject.org
また、Pymatgenの公式ドキュメントは以下である。
pymatgen.org
元素情報の取得
まず、元素に関する情報を取得してみる。今回は、"Si"を例に紹介する。
元素情報は、非常に簡単に取得が可能である。
import pymatgen as mg el = mg.Element('Si') print(el.atomic_mass) #原子量を表示 print(el.melting_point) #融点を表示
>(出力)
28.0855 amu
1687.0 K
このように、非常に簡単に元素情報を取り出すことができる。
取り出せる全ての物性値を以下に示す(少し古い情報なので、今は変更があるかも)。
- Electrical resistivity
- Electronic structure
- Oxidation states
- Reflectivity
- Poissons ratio
- Molar volume
- Van der waals radiu
- ICSD oxidation states
- Bulk modulus
- Mineral hardness
- Mendeleev no
- Atomic no
- Boiling point
- Common oxidation states
- Brinell hardness
- Density of solid
- Thermal conductivity
- Critical temperature
- Name
- Atomic mass
- Coefficient of linear thermal expansion
- Rigidity modulus
- Youngs modulus
- Velocity of sound
- X
- Liquid range
- Atomic radius calculated
- Vickers hardness
- Superconduction temperature
- Melting point
- Refractive index
- Ionic radii
- Atomic radius
上記の物性を一つ一つ取ってくるのは、めんどうなので別の方法がある。
以下のように"el.data"とやると、辞書式で全ての物性値が取り出せる。
なので、for文を使って、全ての特性を簡単に取得することができる。
# 元素名を入れると、物性値が返ってくる関数を作成した。 def get_element_data(name): el = mg.Element(name) for i in el.data: print(i + ' : ' + str(el.data[i])) name='Er' get_element_data(name)
>(出力)
Atomic mass : 167.259
Atomic no : 68
Atomic orbitals : {'1s': -1961.799176, '2p': -302.01827, '2s': -316.310631, '3d': -51.682149, '3p': -63.818655, '3s': -70.310142, '4d': -6.127443, '4f': -0.278577, '4p': -10.819574, '4s': -13.423547, '5p': -0.935202, '5s': -1.616073, '6s': -0.134905}
Atomic radius : 1.75
Atomic radius calculated : 2.26
Boiling point : 3141 K
Brinell hardness : 814 MN m-2
Bulk modulus : 44 GPa
Coefficient of linear thermal expansion : 12.2 x10-6K-1
Common oxidation states : [3]
Critical temperature : no data K
Density of solid : 9066 kg m-3
Electrical resistivity : 86.0 10-8 Ω m
Electronic structure : [Xe].4f12.6s2
ICSD oxidation states : [3]
Ionic radii : {'3': 1.03}
Liquid range : 1371 K
Melting point : 1802 K
Mendeleev no : 22
Mineral hardness : no data
Molar volume : 18.46 cm3
Name : Erbium
Oxidation states : [3]
Poissons ratio : 0.24
Reflectivity : no data %
Refractive index : no data
Rigidity modulus : 28 GPa
Shannon radii : {'3': {'VI': {'': {'crystal_radius': 1.03, 'ionic_radius': 0.89}}, 'VII': {'': {'crystal_radius': 1.085, 'ionic_radius': 0.945}}, 'VIII': {'': {'crystal_radius': 1.144, 'ionic_radius': 1.004}}, 'IX': {'': {'crystal_radius': 1.202, 'ionic_radius': 1.062}}}}
Superconduction temperature : no data K
Thermal conductivity : 15 W m-1 K-1
Van der waals radius : no data
Velocity of sound : 2830 m s-1
Vickers hardness : 589 MN m-2
X : 1.24
Youngs modulus : 70 GPa
Metallic radius : 1.756
iupac_ordering : 36
IUPAC ordering : 36
化合物情報の取得
pymatgenでは、物性値の取得方法は2種類ある(私の知る限り)。
- 組成を入力する
- 条件を指定して習得する
まずは、組成情報を入力して取得する方をやってみる。
組成情報を入力して取得する
取得できる物性情報は、selected_propに書いてある物性値である。
早速、データを取得してみる。
import pymatgen as mg from pymatgen import MPRester #バージョンなどによって、MPResterの位置が異なる場合がある。 # その時は、pymatgenのライブラリを覗きに行ってMPResterの位置を確認にてあげてください。 selected_prop =[ "energy", "energy_per_atom", "volume", "energy", "formation_energy_per_atom", "nsites", "unit_cell_formula", "pretty_formula", "is_hubbard", "elements", "nelements", "e_above_hull", "hubbards", "is_compatible", "spacegroup", "task_ids", "band_gap", "density", "icsd_id", "icsd_ids", #"cif", #表示するとかなり大きくなってしまうので、今回は取得しない。 "total_magnetization", "material_id", "oxide_type", "tags", "elasticity" ] api_key='YOUR_API_KEY' #'SET YOUR API' #自分のAPI keyを入力してください。 with MPRester(api_key=api_key) as m: mater = 'ZnMnO3' #ここに欲しい化合物の組成を書く。 data = m.get_data(mater) #この行で、データを取得している。 for i in data: print('='*30) for j in selected_prop: print(str(j) + ' : ' + str(i[j]))
>(出力)
==============================
energy : -120.56779976
energy_per_atom : -6.028389988
volume : 195.097888591396
energy : -120.56779976
formation_energy_per_atom : -1.7328056347782081
nsites : 20
unit_cell_formula : {'Mn': 4.0, 'Zn': 4.0, 'O': 12.0}
pretty_formula : MnZnO3
is_hubbard : True
elements : ['Mn', 'O', 'Zn']
nelements : 3
e_above_hull : 0.09838204933333117
hubbards : {'Mn': 3.9, 'Zn': 0.0, 'O': 0.0}
is_compatible : True
spacegroup : {'symprec': 0.1, 'source': 'spglib', 'symbol': 'Pnma', 'number': 62, 'point_group': 'mmm', 'crystal_system': 'orthorhombic', 'hall': '-P 2ac 2n'}
task_ids : ['mp-900209', 'mp-772528', 'mp-885520', 'mvc-3995', 'mp-885529', 'mp-1274943', 'mp-1295178', 'mp-1314552', 'mp-1321473', 'mp-1317304', 'mp-1324033', 'mp-1638554', 'mp-1638625', 'mp-1638637', 'mp-1638619', 'mp-1651804', 'mp-1650749', 'mp-1787315', 'mp-900941', 'mp-1979901', 'mp-1981003', 'mp-1980086']
band_gap : 0.11529999999999996
density : 5.731355844333344
icsd_id : None
icsd_ids :
total_magnetization : 2.9994051
material_id : mp-772528
oxide_type : oxide
tags :
elasticity : None
==============================
energy : -27.82799765
energy_per_atom : -5.56559953
volume : 52.26198479368501
energy : -27.82799765
formation_energy_per_atom : -1.2700151767782075
nsites : 5
unit_cell_formula : {'Mn': 1.0, 'Zn': 1.0, 'O': 3.0}
pretty_formula : MnZnO3
is_hubbard : True
elements : ['Mn', 'Zn', 'O']
nelements : 3
e_above_hull : 0.5611725073333318
hubbards : {'Mn': 3.9, 'Zn': 0.0, 'O': 0.0}
is_compatible : True
spacegroup : {'symprec': 0.1, 'source': 'spglib', 'symbol': 'Pm-3m', 'number': 221, 'point_group': 'm-3m', 'crystal_system': 'cubic', 'hall': '-P 4 2 3'}
task_ids : ['mp-1016932', 'mp-1017012', 'mp-1017069', 'mp-1017050', 'mp-1430103', 'mp-1731940', 'mp-1794974', 'mp-1615980']
band_gap : 0.0
density : 5.348894748303294
icsd_id : None
icsd_ids :
total_magnetization : 3.1400626
material_id : mp-1016932
oxide_type : oxide
tags :
elasticity : {'G_Reuss': 61.0, 'G_VRH': 67.0, 'G_Voigt': 73.0, 'G_Voigt_Reuss_Hill': 67.0, 'K_Reuss': 76.0, 'K_VRH': 76.0, 'K_Voigt': 76.0, 'K_Voigt_Reuss_Hill': 76.0, 'elastic_anisotropy': 0.96, 'elastic_tensor': [[225.0, 1.0, 1.0, 0.0, 0.0, 0.0], [1.0, 225.0, 1.0, 0.0, 0.0, 0.0], [1.0, 1.0, 225.0, 0.0, 0.0, 0.0], [0.0, 0.0, 0.0, 47.0, 0.0, 0.0], [0.0, 0.0, 0.0, 0.0, 47.0, 0.0], [0.0, 0.0, 0.0, 0.0, 0.0, 47.0]], 'homogeneous_poisson': 0.16, 'poisson_ratio': 0.16, 'universal_anisotropy': 0.96, 'elastic_tensor_original': [[223.52508206920976, 2.624401788820888, 1.2895446005609252, 0.0, 0.0, 0.0], [0.21993485643860408, 225.78851072542224, 1.2971077031573202, 0.0, 0.0, 0.0], [0.21964388164024937, 2.6278009860233453, 224.5228546177142, 0.0, 0.0, 0.0], [-0.3447858513789858, 0.4339506851651542, -0.02077365489007688, 46.931401255089256, 0.0, 0.0], [-0.21077425914030695, 0.35241040047770134, -0.18308315075587742, 0.0, 46.92248801527998, 0.0], [0.018810794127094042, -0.038505358317629475, -0.007882863514756009, 0.0, 0.0, 46.932124201983065]], 'compliance_tensor': [[4.5, -0.0, -0.0, 0.0, -0.0, 0.0], [-0.0, 4.5, -0.0, 0.0, 0.0, 0.0], [-0.0, -0.0, 4.5, -0.0, -0.0, 0.0], [0.0, -0.0, -0.0, 21.3, -0.0, 0.0], [-0.0, 0.0, -0.0, 0.0, 21.3, -0.0], [0.0, 0.0, 0.0, -0.0, -0.0, 21.3]], 'warnings': , 'nsites': 5}
==============================
energy : -61.20978206
energy_per_atom : -6.120978206
volume : 102.91461477749
energy : -61.20978206
formation_energy_per_atom : -1.8253938527782083
nsites : 10
unit_cell_formula : {'Mn': 2.0, 'Zn': 2.0, 'O': 6.0}
pretty_formula : MnZnO3
is_hubbard : True
elements : ['Mn', 'O', 'Zn']
nelements : 3
e_above_hull : 0.005793831333330779
hubbards : {'Mn': 3.9, 'Zn': 0.0, 'O': 0.0}
is_compatible : True
spacegroup : {'symprec': 0.1, 'source': 'spglib', 'symbol': 'R-3', 'number': 148, 'point_group': '-3', 'crystal_system': 'trigonal', 'hall': '-R 3'}
task_ids : ['mp-770583', 'mp-803358', 'mp-811615', 'mp-754318', 'mp-830632', 'mp-1298159', 'mp-1292723', 'mp-1284960', 'mp-1276065', 'mp-1293892', 'mp-1298358', 'mp-1297282', 'mp-1296202', 'mp-1298447', 'mp-1296247', 'mp-1286252', 'mp-1286337', 'mp-1281904', 'mp-1291948', 'mp-1295441', 'mp-1279918', 'mp-1387979', 'mp-1752477', 'mp-1773640', 'mp-831650', 'mp-812278', 'mp-1621284']
band_gap : 1.7281
density : 5.432539520324601
icsd_id : None
icsd_ids :
total_magnetization : 2.99992435
material_id : mp-754318
oxide_type : oxide
tags : []
elasticity : None
こんな感じでデータが取得できる。
複数の化合物についてデータが取得されているが、これは入力した組成に多形が存在していることを意味している。
条件を指定して、取得する方法
含む元素を指定して化合物データを抽出する
La-Li-O-Zr4元系の化合物を取得のデータを取得してみる。
import pandas as pd #Material IDを取得して、テキストファイルに書き込む関数。 #この関数では、元素系を指定してMaterial IDを取得してきている。 def get_mp_ids(element_list,criteria,properties,output): properties=list(set(properties)) df=pd.DataFrame(columns=properties) #取得したデータをcsvにまとめたいため、pandasを利用する。 with open(output,'w') as w: with MPRester(api_key) as m: for i in [element_list]: data = m.query(criteria=criteria,properties=properties) #ここで条件と取得したい特性を指定している for d in data: #print(d) df.loc[d['material_id']]=list(d.values()) return df properties=[ "material_id", #"energy_per_atom", #"volume", #"energy", "formation_energy_per_atom", #"nsites", #"unit_cell_formula", "pretty_formula", #"is_hubbard", #"elements", #"nelements", "e_above_hull", #"hubbards", #"spacegroup", #"task_ids", "band_gap", #"density", #"icsd_id", #"icsd_ids", #"cif", #"total_magnetization", #"oxide_type", #"tags", #"elasticity" ] output = ':La-Li-O-Zr.txt' element_list = ['La','Li','O','Zr'] criteria={"elements": {'$in':[['La','Li','O','Zr' ]]}} #La-Li-O-Zr系の化合物を取得 #['La','Li','O','Zr' ]にすると、La,Li,O,Zrそれぞれを含む化合物(7万件くらい)をとってくるので注意 # 書き方の参考: https://pymatgen.org/pymatgen.ext.matproj.html df = get_mp_ids(element_list,criteria,properties,output) df

また、criteriaを"*-Li-O-Zr"とすると、Li-O-Zrを同時に含む全ての4元系化合物を取得することができる。
criteria="*-Li-O-Zr" #Li-O-Zrを同時に含む化合物を全て取得する df = get_mp_ids(element_list,criteria,properties,output) df

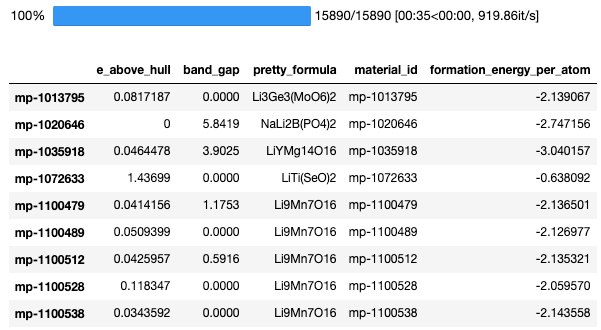
例:Li-O系化合物の取得
criteria={"elements": {"$all": ['Li','O']}}とすることで、LiとOを含む全ての化合物を取得することができる。
properties=[
"material_id",
#"energy_per_atom",
#"volume",
#"energy",
"formation_energy_per_atom",
#"nsites",
#"unit_cell_formula",
"pretty_formula",
#"is_hubbard",
#"elements",
#"nelements",
"e_above_hull",
#"hubbards",
#"spacegroup",
#"task_ids",
"band_gap",
#"density",
#"icsd_id",
#"icsd_ids",
#"cif",
#"total_magnetization",
#"oxide_type",
#"tags",
#"elasticity"
]
output = 'Li-O.txt'
criteria={"elements": {"$all": ['Li','O']}} #Liと0を同時に含む全ての化合物を取得する
df = get_mp_ids(element_list,criteria,properties,output)
df
物性値で条件指定
criteria={"elements": {"$all": ['Li','O']},"band_gap":{"$gt":1}}とすると、 LiとOを同時に含むかつ、バンドギャップが1eV以上である化合物を取得することができる。
もちろん、他の物性値でも条件指定は可能である。
また、gtはgreater thanの意味である。 これらの数値条件の指定方法を下の表に示す。

output = 'Li-O_bandgap_gt1.txt' criteria={"elements": {"$all": ['Li','O']},"band_gap":{"$gt":1}} #バンドギャップが1eV以上であり、かつLiと0を同時に含む全ての化合物を取得 df = get_mp_ids(element_list,criteria,properties,output) df

まとめ
Pymatgenを使いこなすことができれば、このような巨大なデータベースから簡単にデータ取得ができるようになる。 また、化合物の解析も簡単に行えるようになる。 また、時間があるときに、Pymatgenの使い方を書いていこうと思う。